正则表达式( , regex)是一个重要且实用的概念,我时常提起却从未细谈。一怕能力不够说不清楚反而会误导人grep正则表达式,二是已经有无数前人撰文介绍。考虑日常用到的grep,sed,awk里经常需要用到正则表达式,于是开一个小系列,介绍如何在grep,sed,awk中适用正则。
什么是正则表达式
正则表达式( )的概念,最初来自于20世纪40年代的两位神经学家( , Pitts)研究神经元时提出的想法。后来数学家 在代数学中正式描述了这种被他称之为“正则集合”的模型。并且,他还发明了一套简洁的方法表示正则集合,也就是正则表达式。
目前最快速的文本搜索工具grep就内置了正则表达式。grep起源于Unix中的ed编辑器的一条命令g//p, 读作“ Print”,即运用正则表达式的全局输出。由于这个功能太过实用,于是就从ed中独立出来,演变成了grep以及扩展版本的egrep。都知道grep因为有正则表达式所以很强大,但是正则表达式为何如此强大呢?
正则表达式的强大之处在于它是一套语法,分为两个部分,元字符()和普通文本字符( text )。
以语言类比grep正则表达式,“我爱正则表达式”这句话可以抽象成“主谓宾”结构,主语是"我",谓语是"爱",宾语是“正则表达式”。这种结构还适用于其他语言,比如说英语就是"I love ". 这种语法结构就是元字符,而构成句子的语言就是普通文字字符。
元字符一览
正则表达式有很多流派,不同流派之间的差异在于对元字符的支持程度。以下的元字符适用于GNU版本的grep, sed, awk. mac自带的是BSD版本。
匹配单个字符的元字符:
元字符匹配对象
匹配单个任意字符
[...]
匹配单个列出的字符
[^...]
匹配单个未列出的字符
char
转义元字符成普通字符
提供技术功能的元字符
元字符匹配对象
?
匹配0或1次
匹配0到n次
至少一次,最多不限
{min,max}
至少min次, 最多max次
匹配位置的元字符
元字符匹配对象
匹配一行的开头
匹配一行的结尾
其他元字符
元字符匹配对象
竖线
匹配任意分割的表达式
(...)
限定多选结构的范围,标注量词的作用范围,为反向引用捕获元素
1, 2
反向引用元素
小字:awk, grep, sed支持perl的正则系统,而perl的正则表达式是及其强大的,后续可能会说到。
在grep中使用正则表达式
grep的强大之处它所做的事情就只有在文本搜索”正则表达式“定义的模式(),如果找到就打印出来。可以使用man egrep查看所支持的参数。
egrep [options] pattern [file]
egrep [options] [-e pattern]... [-f FILE]... [FILE...]
# 参数参数
-e PATTERN: 定义多个模式
-f FILE: 从文本中读取模式
-w: 匹配整个单词
-v: 反向匹配
-i: 忽略大小写
-x: 仅仅选择整行匹配结果
-c: 计数
-n: 输出表明行号
-A/-B NUM: 同时输出后/前几行
注: grep有基础和扩展两个模式,基础模式支持的元字符较少,而egrep表示扩展的grep,支持的元字符较多。
检查测序结果的接头
在分析的质控阶段,需要检查给出的结果是否含有接头()。除了, 还能用grep检测是否有接头序列。
你可以去的官网根据公司测序的查找对应的接头:
或者是直接看的配置文件中检测的接头
# 下载测试数据
fastq-dump -X 10000 SRR1553606 --split-files
然后用部分的接头对fastq文件进行搜索
egrep -B1 'TCGGAA' SRR1553606_1.fastq
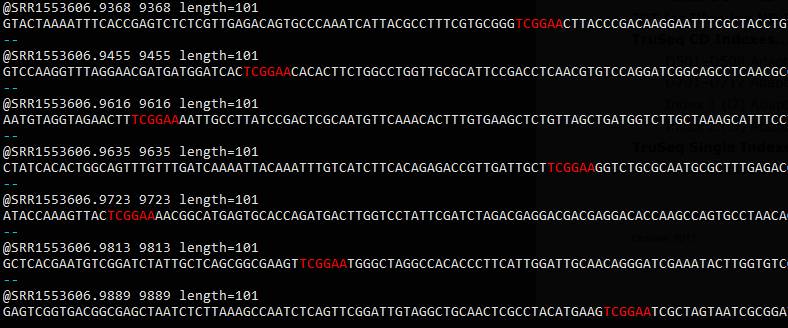
被找到的序列并非出现最开头,你可能希望是开头有几个其他碱基,然后跟着接头序列.
egrep -B1 '^[ATGC]{0,5}TCGGAA' SRR1553606_1.fastq
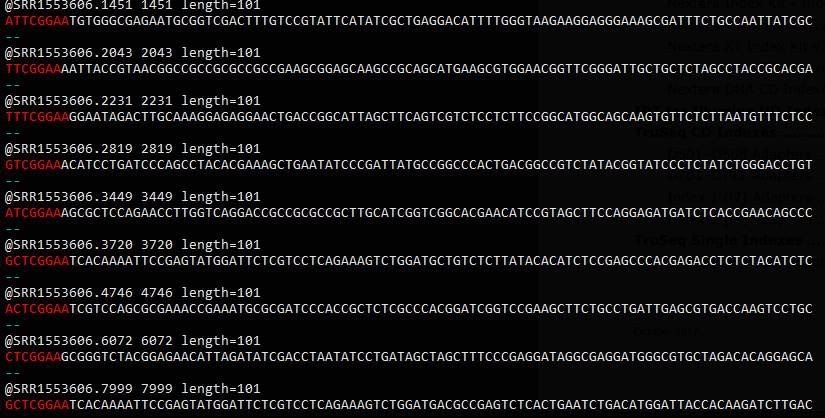
基因搜索
一般而言,大家都回去TAIR上查找基因的区间。但是实际上我们可以先下载好拟南芥参考基因组及其注释文件,然后直接用正则表达式确定区间,用提取序列进行了。
# 下载序列
wget -c -4 -q http://www.arabidopsis.org/download_files/Genes/TAIR10_genome_release/TAIR10_chromosome_files/TAIR10_chr_all.fas
# 下载GFF文件
wget -c -4 -q http://www.arabidopsis.org/download_files/Genes/TAIR10_genome_release/TAIR10_gff3/TAIR10_GFF3_genes.gff
# 基因的信息
wget http://www.arabidopsis.org/download_files/Genes/TAIR10_genome_release/gene_description_20131231.txt.gz
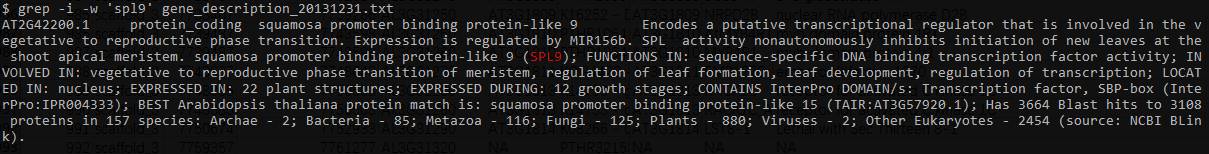
在注释文件中查找某一个基因如SPL9, 并获取他的位置信息.
grep -i -w 'spl9' gene_description_20131231.txt
grep -e 'AT2G42200.1' TAIR10_GFF3_genes.gff | grep 'mRNA'
当然以上命令可以连成管道, 并且和合用,就可用直接获取DNA序列。
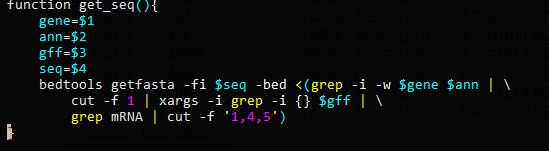
bedtools getfasta -fi TAIR10.fa -bed
<(grep -i -w 'spl9' gene_description_20131231.txt |
cut -f 1 | xargs -i grep -i {} TAIR10_GFF3_genes.gff | grep mRNA | cut -f '1,4,5')
你可以将其定义成一个函数,存放在配置文件中, 随后就能进行调用该函数。
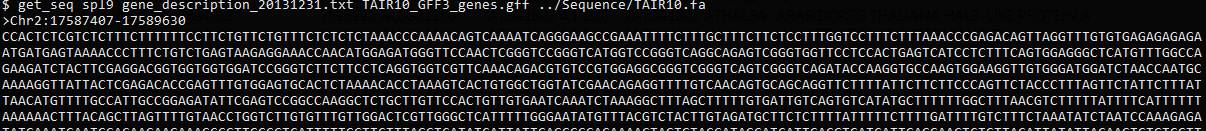
这样子就不需要去TAIR才能找基因序列了,而且速度更快,没有网络延迟哦
限时特惠:本站持续每日更新海量各大内部创业课程,一年会员仅需要98元,全站资源免费下载
点击查看详情
站长微信:Jiucxh




